 数据资源
计算分析
标准规范
数据网络
数据资源
计算分析
标准规范
数据网络
| 名称 | deNOPA |
|---|---|
| 类别 | 基因组组装与注释 |
| 版本号 | v1.0.2 |
| 开发者 | 徐炳祥、李小丽、高晓萌、贾艳、刘静、李飞飞、张治华 |
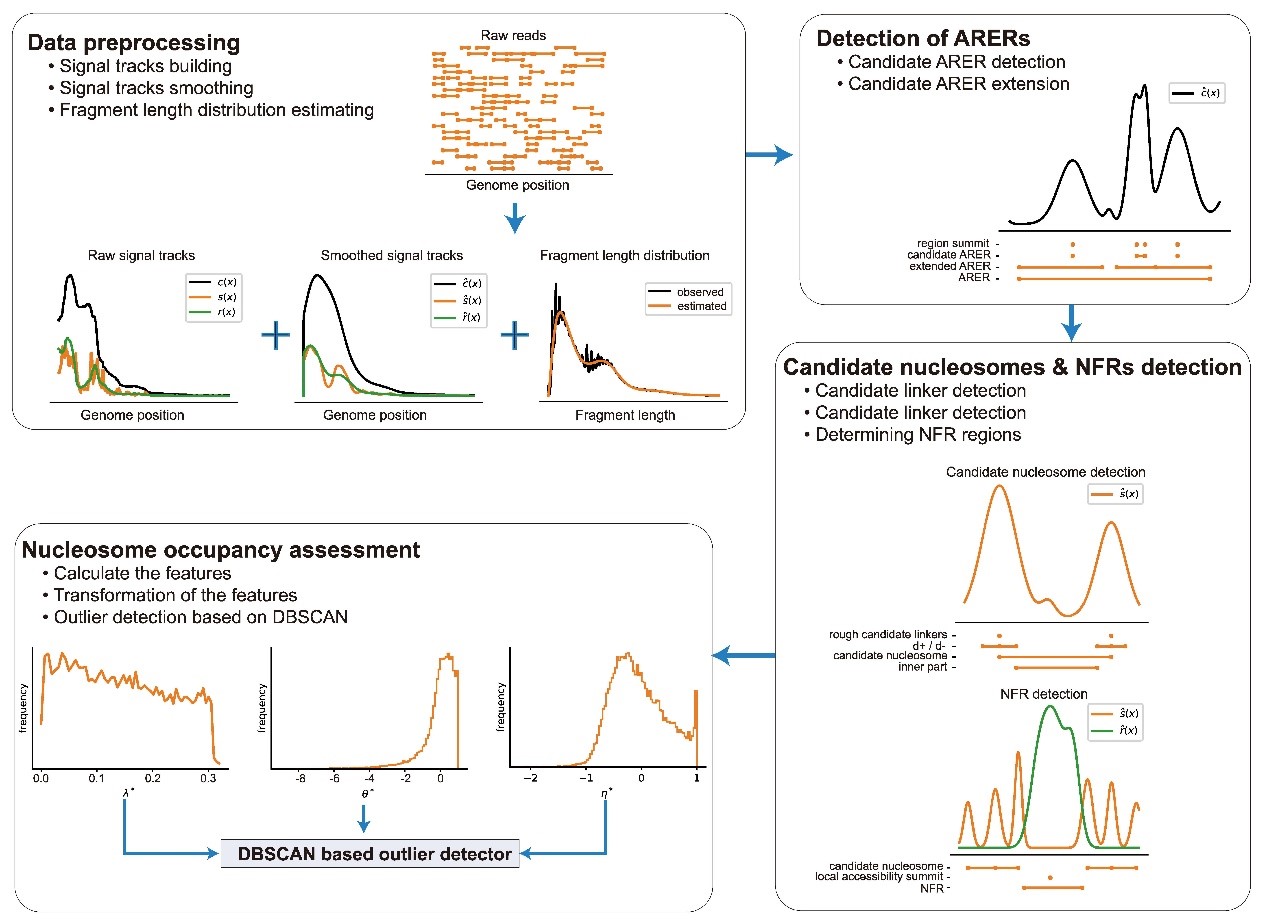
| 描述 | 核小体动态和排列是染色质结构和调控的基础,影响几乎所有的细胞核生物学过程。ATAC-seq以其对样品要求低、操作简便高效的特点已迅速被作为在群体和单细胞水平上分析染色质开放性的主要工具;然而,用ATAC-seq描绘核小体排布本身仍然是一个挑战。此处我们开发了一个名为的deNOPA的ATAC-seq分析工具包,用于预测核小体的位置。评估显示,deNOPA在处理超稀疏ATAC-seq数据(例如每碱基对不超过0.5个片段)时优于现有最佳工具。deNOPA的显着性能主要源于短片段的利用,这些片段占ATAC-seq文库中测序读长的近一半,而通常被现有核小体定位工具丢弃。然而,我们发现这些短片段富集核小体位置信息。我们的deNOPA提供了一个强大的工具,可以用超稀疏ATAC-seq数据来分析染色质在核小体位置水平的动态。 |
| 下载地址 | https://gitee.com/bxxu/denopa |
| 文章发布 | https://pubmed.ncbi.nlm.nih.gov/34875002/ |
| 引用次数 | 5 |
| 图片描述 |
|

北京市朝阳区北辰西路一号院104号 中国北京,100101 | 86-10-84097216
版权所有 © 国家生物信息中心 2025, 京ICP备 10050270号-13